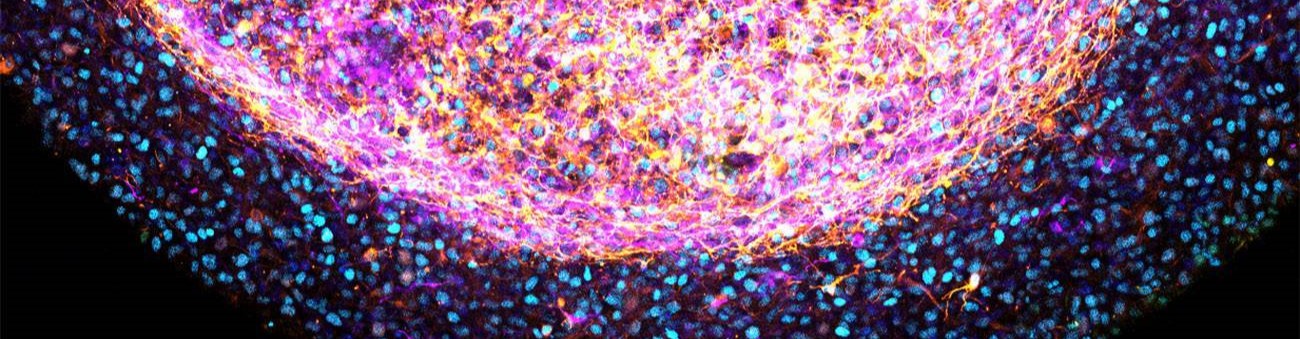
n grupo de científicos en el Centro Nacional de Análisis Genómico (CNAG-CRG) del Centro de Regulación Genómica (CRG) en Barcelona, liderado por Holger Heyn, responsable del equipo de Genómica de Células Individuales en el CNAG-CRG y autor sénior del este trabajo, ha desarrollado una nueva herramienta computacional, basada en la teoría matemática de gráficas, para deducir redes de regulación globales a gran escala, tanto de órganos sanos como patológicos de enfermedades como diabetes o Alzheimer. Los investigadores pudieron precisar la relevancia génica en la función del órgano y posibles promotores de enfermedades, según los resultados que se han publicado en el último número de Genome Biology.
Según Giovanni Iacono, investigador postdoctoral senior en el CNAG-CRG y primer autor del estudio las herramientas de transcriptómica de células individuales, desarrolladas con anterioridad, resultaron muy útiles para descubrir tipos de células desconocidos. “Las herramientas nos permitieron describir nuevos tipos y subtipos de células, junto con sus roles biológicos únicos y relaciones jerárquicas”.
Los investigadores del CNAG-CRG procesaron conjuntos de datos de 10.000 células para deducir las redes de regulación que impulsan la formación del fenotipo de la célula y sus respectivas funciones. La herramienta se ha aplicado en el estudio de la diabetes tipo 2 y del Alzheimer y se descubrieron cambios funcionales relevantes para la enfermedad.
Una nueva técnica visualiza cómo interactúan los genes para formar órganos y tejidos